最新の研究
- 2019.10.01
- 田中・吉田研究室
植物細胞のモデル系、単細胞原始紅藻シゾンにおける核ゲノム複数箇所の改変技術を確立
【研究背景】
生物を構成するメカニズムは非常に複雑であり、まだまだ未解明な部分が多く残されています。それらを理解するためには、より単純な系に落とし込み、基本原理を見いだすことが効率の良い戦略です。そこで私たちは研究の材料として、モデル生物である単細胞紅藻Cyanidioschyzon merolae(通称シゾン)に着目しています。シゾンは様々な証拠により、最も原始的な真核細胞の一つであると考えられています[1]。また、核、オルガネラのゲノム配列が全て決定されており、それらの情報に基づいて、ゲノム改変などの分子遺伝学的手法を用いることで、遺伝子の機能解析を行うことが可能です。これまでもシゾンを用いて様々な研究が展開されてきました。
ゲノム改変とは、外来のDNAを細胞内に導入して特定の遺伝子に変異を施すものですが、ゲノム改変には、外来DNAが導入された組換え体をその他の細胞から区別するための選択マーカー遺伝子を用いる必要があります。このため、特定の選択マーカー遺伝子を用いて作成した組換え体のゲノムには、用いた選択マーカー遺伝子が残され、同じマーカー遺伝子はさらなるゲノム改変には用いることができません。シゾン核ゲノムでは、相同組換えに基づいたゲノム改変の技術が確立されています。しかし、これまでは選択マーカー遺伝子の数が限られているためにゲノムの複数箇所の改変を行うことは困難でした。現在までに利用可能な選択マーカーとしては、2008年に報告されたウラシル合成マーカー遺伝子(URA5.3)[2]、および近年報告されたクロラムフェニコール耐性遺伝子(CAT)[3]のみです。そこで私たちは本研究で、URA5.3遺伝子を繰り返し利用することでゲノムの複数箇所を改変する技術を開発しました。
【研究成果】
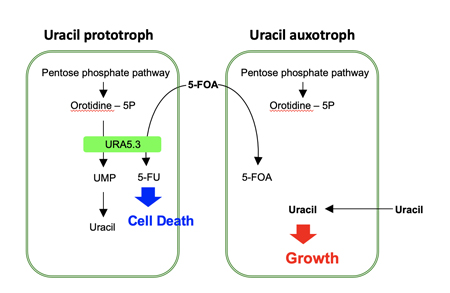
選択マーカー遺伝子を繰り返し利用するためには、一度導入した選択マーカー遺伝子をゲノム上から取り除く必要があります。そこで、私たちは、URA5.3遺伝子がウラシル合成に必要な基質となるウリジン5'-リン酸(UMP)の生合成に働くと同時に、ウラシル生合成系の前駆体、オロチジン5'-リン酸のアナログである5-フルオロオロト酸(5-FOA)を細胞毒性のある5-フルオロウリジン(5-FU)に変換することに着目しました(Fig. 1)。この性質により、5-FOAの存在下ではURA5.3遺伝子を持たない株を選抜することが可能となります。
Fig. 1 選択マーカー遺伝子URA5.3の特長
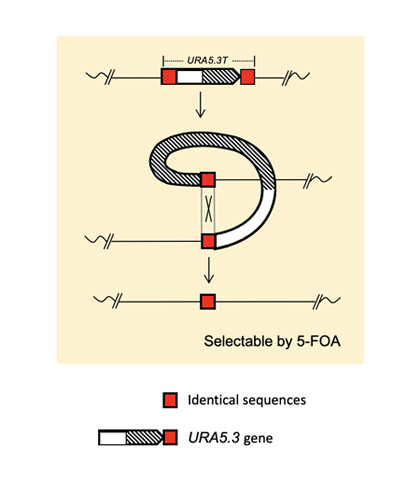
本研究では、一旦導入したURA5.3遺伝子を相同組換えにより欠失させるため、マーカー遺伝子の5'側と3'側に約500 bpのリピート配列を配置した改変URA5.3遺伝子(URA5.3-T)を作製しました[4](Fig. 2)。5-FOA存在下では、URA5.3遺伝子が細胞毒性のある5-FUを産生してしまうため、URA5.3遺伝子を失った細胞を選択することができます。
Fig. 2 URA5.3-T欠失株を5-FOAにより選択する概念図
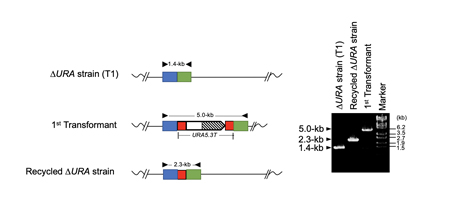
次に、実際にFig. 2のような相同組換えが起きるのかどうか検証する実験を行いました。まず、URA5.3遺伝子が欠失したウラシル要求性の宿主株(T1)[5]に対して、URA5.3-T遺伝子を組み込んだDNA断片を導入し、ウラシル非要求性により形質転換株(transformant)を選抜しました。その後、得られた形質転換体を5-FOA含有固形培地上で選抜しました。その結果、予想通りURA5.3遺伝子が欠失した形質転換体(Recycled DURA strain)を取得することができました。図3は、これらの株から抽出したゲノムDNAをPCR法により解析した結果であり、予想通りの遺伝子組換えが起きていることが確認されました(Fig. 3)。
Fig. 3 ゲノム改変を確認するためのPCR実験の結果
【今後の展開】
生物は類似の機能を持つ遺伝子を複数持つことが多いことが知られています。このため、遺伝子の機能解析を行う際に、機能を知りたい特定の遺伝子を破壊しても、他の同様の機能を持つ遺伝子により機能が相補され、変異による形質の変化が観察できないことがあります。そのような時には、同様の機能を持つ遺伝子を多重に破壊することで機能解析を進めることが必要です。今回の研究成果は、シゾンにおいて、このような遺伝子機能の解析を進めることを可能にしたものです。ゲノム改変によるバイオマスや有用物質生産株の構築においても[6,7]、複数の遺伝子領域を同時に改変する技術は重要です。本研究で開発された多重遺伝子変異系を用いることで、有用物質を合成するための遺伝子を多重に導入することができるため、有用物質生産の効率化につながることが期待されます。
参考文献
- [1] Kuroiwa T. (1998) The primitive red algae Cyanidium caldarium and Cyanidioschyzon merolae as model system for investigating the dividing apparatus of mitochondria and plastids. Bioessays, 20, 344-354.
- [2] Ohnuma M., Yokoyama T., Inouye T., Sekine Y. and Tanaka K. (2008) Polyethylene glycol (PEG)-mediated transient gene expression in a red alga, Cyanidioschyzon merolae 10D. Plant Cell Physiol., 49, 117-120.
- [3] Fujiwara T., Ohnuma M., Kuroiwa T., Ohbayashi R., Hirooka S. and Miyagisima S.Y. (2017) Development of a double nuclear gene-targeting method by two-step transformation based on a newly established chloramphenicol-selection system in the red alga Cyanidioschyzon merolae. Front. Plant Sci., 8, 343.
- [4] Takemura T., Imamura S., Kobayashi Y. and Tanaka K. (2018) Construction a selectable marker recycling system and the use in epitope tagging of multiple nuclear genes in the unicellular red alga Cyanidioschyzon merolae. Plant Cell Physiol., 59, 2308-2316.
- [5] Taki K., Sone T., Kobayashi Y., Watanabe S., Imamura S. and Tanaka K. (2015) Construction of a URA5.3 deletion strain of the unicellular red alga Cyanidioschyzon merolae: A backgroundless host strain for transformation experiments. J. Gen. Appl. Microbiol., 61, 211-214.
- [6] Yamaguchi S., Kawada Y., Yuge H., Tanaka K. and Imamura S. (2017) Development of new carbon resource: Production of important chemicals from algal residue. Sci. Reports, 7, 855.
- [7] Takemura T., Imamura S. and Tanaka K. (2019) Identification of a chloroplast fatty acid exporter protein, CmFAX1, and triacylglycerol accumulation by its overexpression in the unicellular red alga Cyanidioschyzon merolae. Algal Res., 38, 101396.