最新の研究
- 2021.07.01
- 田中・吉田研究室
シアノバクテリア系統内で起きた染色体DNA複製開始機構の進化と染色体倍数化
染色体DNAの複製は遺伝情報を継承する上で最も重要なプロセスの一つである。染色体DNA複製制御機構は開始段階の制御が主であり、生物種間で非常によく保存されている。バクテリアはDnaA、真核生物、アーキアはORCという複製開始因子により制御されており、この因子は現在ゲノム情報が公開されているほぼすべての生物に保存されていることから、複製開始機構は生命が多様化する以前に獲得した共通の機構であり、特にバクテリアではDnaA依存的な複製機構はドグマ化されてきた背景がある。しかし一方で、近年ゲノム解読が進み、興味深いことに細胞内共生しているバクテリアの多くがdnaA遺伝子を失っていることが分かってきた。さらに、バクテリアが共生し誕生した葉緑体やミトコンドリアのようなオルガネラゲノム(さらに核ゲノム)にもdnaA遺伝子は保存されていなことから、現存のオルガネラはバクテリア型(DnaA依存型)の複製開始機構ではないことが示唆される。しかし、いつ、どのような機構に進化したのかは全く分かっていなかった。我々は以前、シアノバクテリアの一部はdnaA遺伝子が必須ではなく、DnaA非依存の複製機構へと進化している可能性を示した1)。今回我々はさらに系統的に異なるシアノバクテリアを用いて、この進化はシアノバクテリア系統内で複数回独立に起き、細胞内共生以前にDnaA非依存的な複製開始機構を獲得していたことを報告したので紹介する2)。
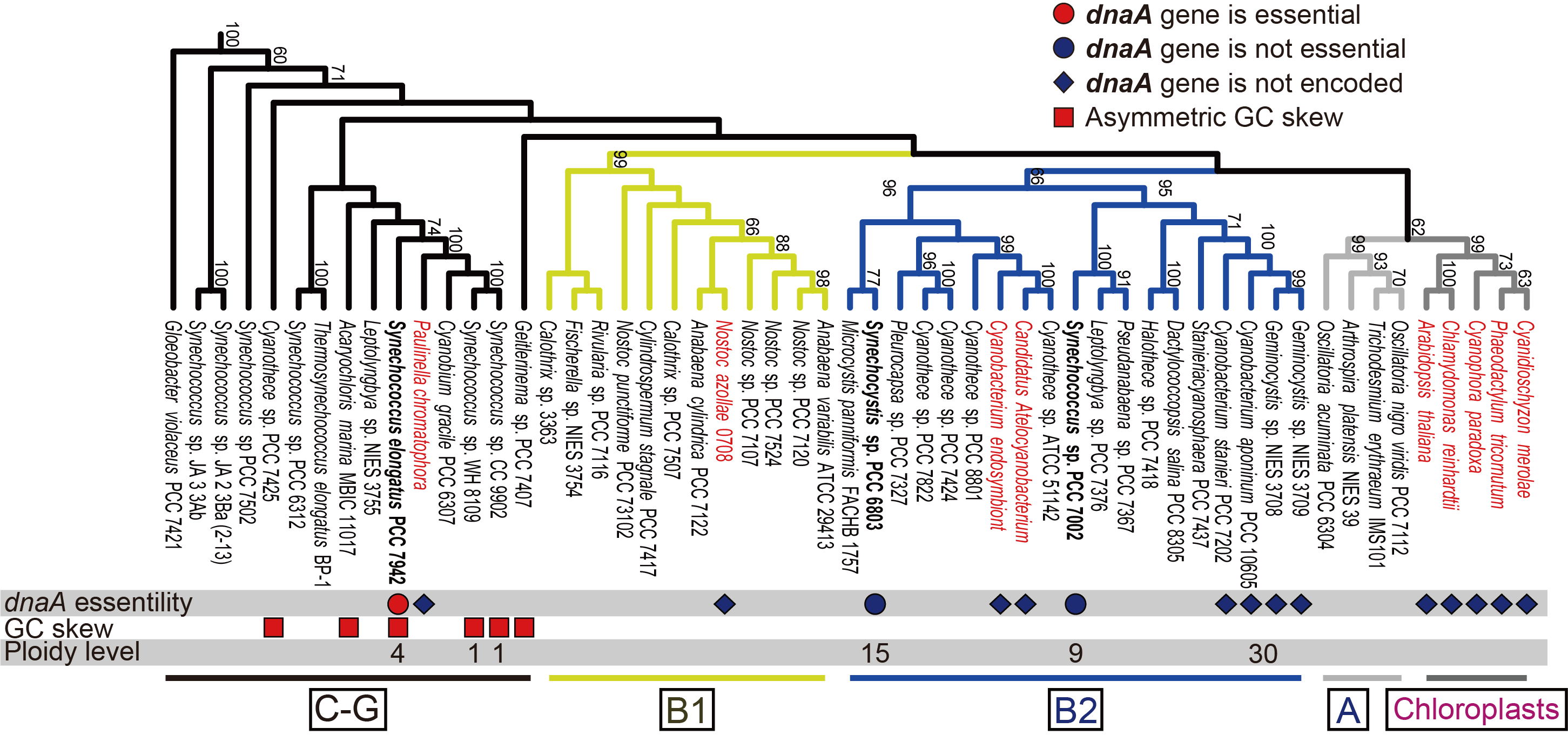
まず、我々はゲノムが完全解読されているシアノバクテリアにおいて、ゲノム構造やdnaA遺伝子の有無を系統的に解析した。その結果、非常に興味深いことにすでにdnaA遺伝子を失ったFree-livingのシアノバクテリアが存在していることが明らかとなった(図1; Cyanobacterium stanieri PCC 7202、Cyanobacterium sp. PCC 10605、Geminocystis sp. PCC 6308、Geminocystis NIES-3708と3709)。系統関係から考えると、共生しているシアノバクテリアや葉緑体とは独立してdnaA遺伝子を失ったことがわかる(図1)。これまでdnaA遺伝子を失ったFree-livingのバクテリアは報告がなく、共生以前にすでにdnaA遺伝子を失う方向に進化しうることがわかった。
 |
|||
| 図1. | シアノバクテリアと葉緑体ゲノムにおけるdnaAの系統的分布 | ||
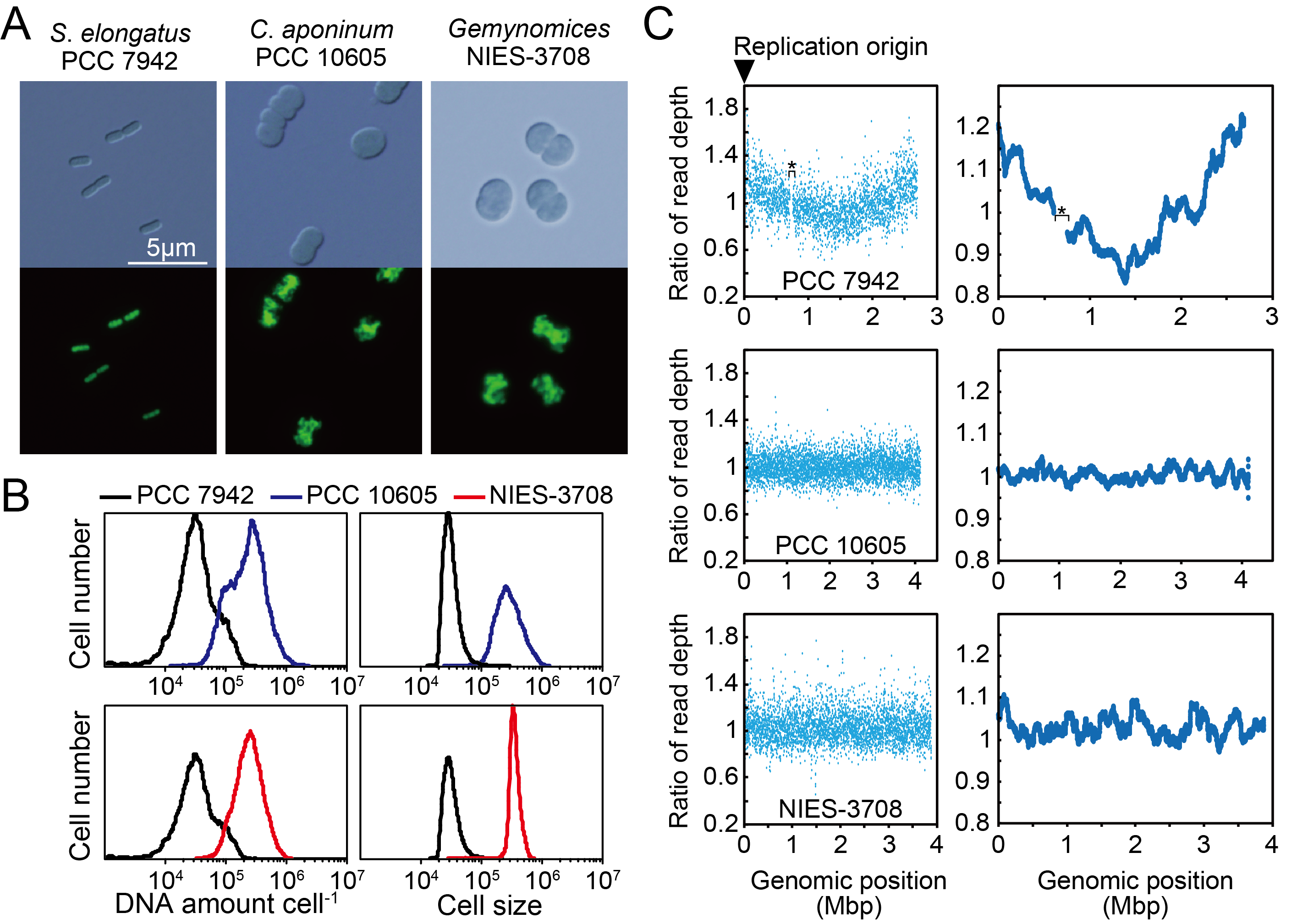
次にこれらdnaA遺伝子を失った2種のシアノバクテリアにおいて、形態的な観察を行ったところ、DnaA依存だとわかっているS. elongatusと比べ、細胞サイズが約8倍程度大きいことが顕微鏡観察やフローサイトメーターによる解析からわかってきた(図2A、B)。さらに、染色体のコピー数を調べた結果、細胞サイズと同様にS. elongatusに比べ8倍程度多いことがわかり、1細胞あたり平均で30コピー程度の染色体を保持している倍数体であることが明らかとなった(図2A、B)。
DnaA依存のバクテリアはS. elongatusのようにゲノム上のある一点から複製が開始することが分かっている(図2C)。ではdnaAがないシアノバクテリアは一体どこから複製を開始しているのか?この疑問を調べるために次世代シーケンサーによってゲノムワイドに開始点の探索を行った。その結果、S. elongatusのような特異的なピーク(複製開始点)は検出できなかった(図2C)。理由はいくつか考えられ、コピー数が多いため、一部の染色体しか複製していない場合は複製していな染色体との差が小さく検出できないか、もしくは複製開始点が複数存在する可能性も考えられるが、現状では詳しい複製開始点の同定には至っていない。
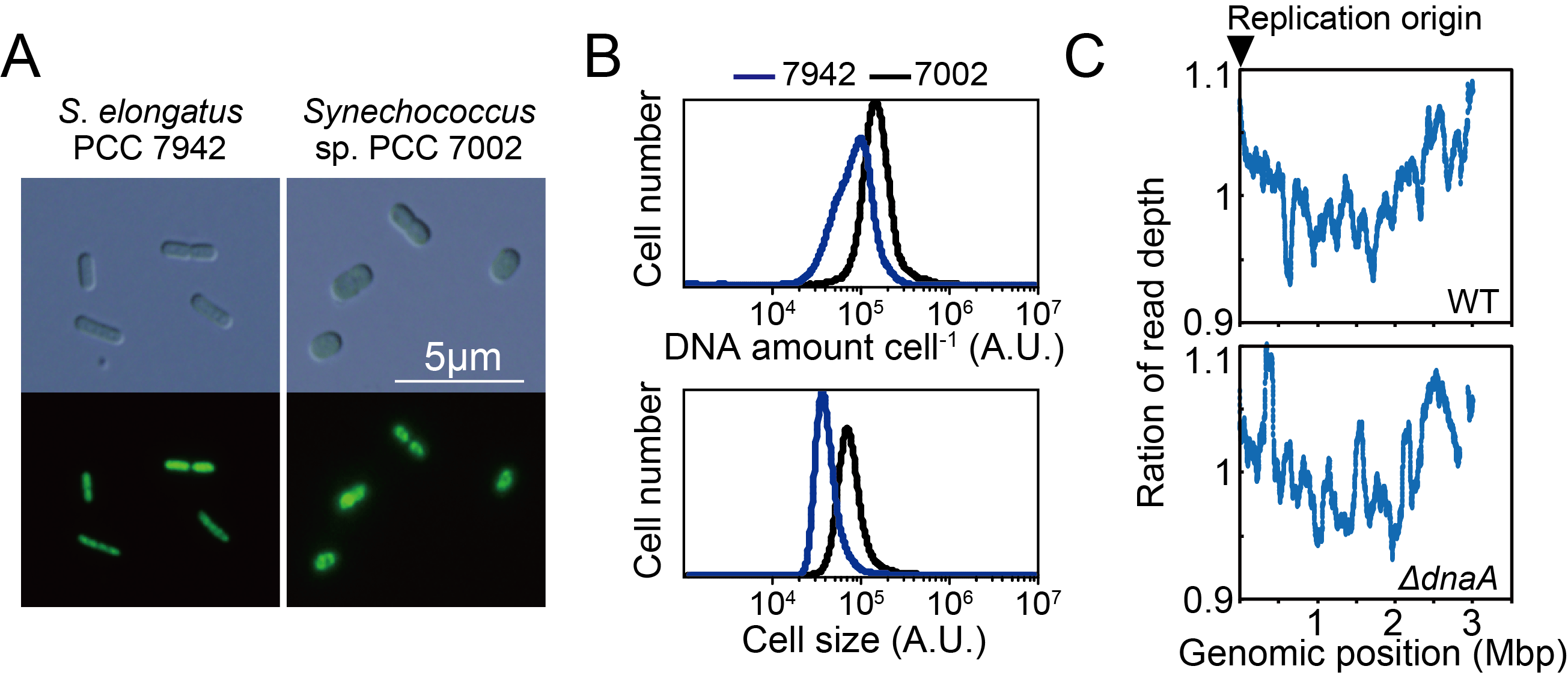
次にどの段階でDnaA非依存へと進化したのかを検証するため、系統的に近くdnaA遺伝子をコードするSynechococcus sp. PCC 7002において(図1)、dnaA遺伝子の必須性や複製開始点を解析した。その結果、PCC 7002においてdnaA遺伝子欠損しても生育、複製活性に影響はないことがわかった。一方で興味深いことに、複製開始点を次世代シーケンサーで解析した結果、特定の複製開始点から複製が始まることがわかった(図3C)。またdnaA遺伝子欠損株でも同様の複製開始点が検出されたことから、PCC 7002はすでにdnaA遺伝子は必須ではなくDnaA非依存的な複製開始機構であることが示された。
以上のように、シアノバクテリアにはDnaA依存する種と依存しない種が存在し、DnaA非依存への複製開始機構の進化はシアノバクテリアの系統内で複数回独立に起きたことが明らかになった(図1)。さらに、DnaAに依存している種に比べ、DnaA非依存へと進化した種は1細胞あたりの染色体コピー数が増加している傾向にあることもわかってきた(図1)。今後はDnaAに依存しない複製開始機構だけでなはなく、複製開始機構の進化と染色体倍数性との因果関係を明らかにし、共生進化の過程でどのように複製開始機構が進化したのかを明らかにすることを目指す。
Reference
| 1. | Katayama, T., Ozaki, S., Keyamura, K., and Fujimitsu, K. (2010). Regulation of the replication cycle: conserved and diverse regulatory systems for DnaA and oriC. Nature Reviews Microbiology 8, 163-170. |
| 2. | Ohbayashi, R., Watanabe, S., Ehira, S., Kanesaki, Y., Chibazakura, T., and Yoshikawa, H. (2016). Diversification of DnaA dependency for DNA replication in cyanobacterial evolution. The ISME journal 10, 1113-1121. |
| 3. | Ohbayashi R, Hirooka S, Onuma R, Kanesaki Y, Hirose Y, Kobayashi Y, Fujiwara T, Furusawa C, and Miyagishima SY. (2020) Evolutionary changes in DnaA-independent chromosomal replication in cyanobacteria. Frontiers in Microbiology. 11:786. |