最新の研究
- 2015.04.28
ゲノム転写制御システムの解明に向けた転写制御因子の階層性の解析
1.はじめに
生物はゲノムDNAから選択的に遺伝子を発現させることで、環境の変化に適応しています。遺伝情報の発現の調節は、主にDNAからRNAを合成する転写の段階で行われています。転写の制御はRNA合成装置のRNAポリメラーゼにシグマ因子および転写因子が相互作用することで行われます(参考文献1)。近年のシークエンサー技術の発展により様々な生物のゲノムが明らかとなってきており、その生物がどのような遺伝子セットを持っているかは推測することが容易となってきました。その一方で、その遺伝子セットをどのように利用しているかという制御についてはゲノム情報のみからは明らかとすることができず、そのゲノム転写の制御システムを解明することは現在の先端的研究課題の一つとなっております。
2.転写制御因子の直接的な標的遺伝子の同定の必要性
従来の転写制御の解析手法に、転写制御因子の欠損株を作製して遺伝子発現を親株と比較することで、その影響を観察するという方法があります。現在ではゲノム情報を扱うことができるため、全RNAを観察するトランスクリプトーム解析や全タンパク質を観察するプロテオーム解析などが確立されています。これらの手法は細胞全体を対象とした網羅的な解析手法であり、対象としている転写制御因子が細胞に与える影響の全てを反映した結果を観察できる反面、直接的な影響と間接的な影響を区別することができません。さらに、これまでの研究により、転写制御因子同士で制御ネットワークを形成していることも分かってきており、転写制御因子には階層性が存在することが報告されています(参考文献2)。そのため、ゲノム転写制御システムの分子機構を理解するためには、転写制御因子の直接的な標的遺伝子を同定する手法が必要です。
3.Genomic SELEX法を用いた研究成果
そこで私たちは、転写制御因子のゲノム上の標的を同定するGenomic SELEX法を開発しました(参考文献3)。この手法は、試験管内において精製したタンパク質と断片化されたゲノムDNAのライブラリーを混合し反応させた後、タンパク質と相互作用したDNA断片を増幅し、そのDNA配列を解析することで、タンパク質のゲノム上の結合領域、つまり標的遺伝子を同定する、というものです(図1)。
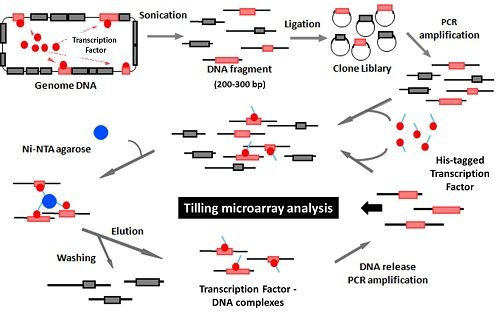
図1.Genomic SELEX法の概略
当初は精製したDNA配列をシークエンスで一つずつ決定していましたが、現在ではタイリングアレイを用いてゲノムレベルで網羅的に解析することが可能となりました(参考文献4)。この技術を用いて、私たちは近年、大腸菌RNAポリメラーゼのゲノム上の結合領域を同定することにも成功しました(図2、参考文献5、6)。
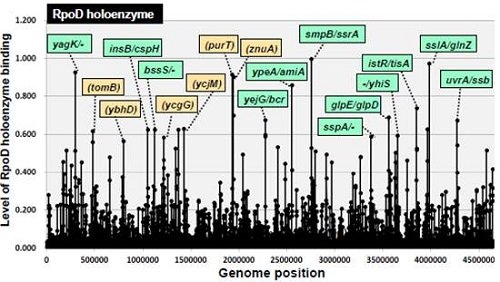
図2.大腸菌RNA polymerase (Sigma70)のゲノム上の分布
この実験はRNAポリメラーゼおよびシグマ因子の一つであるSigma70とゲノムDNAのみの反応で行われたため、同定されたDNA結合領域は'RNAポリメラーゼのホロ酵素単体で認識されるプロモーター'であるということがいえます。さらに、今回の結果をこれまでに報告されている細胞内において同定されたプロモーターと比較することにより、RNAポリメラーゼのホロ酵素に加えて転写因子などのなんらかの他因子による'誘導が必要なプロモーター'とを区別することができました。そこで私たちは前者のプロモーターを'Constitutive promoter'、後者のプロモーターを'Inducible promoter'と区別することを提案しました。このように、Genomic SELEX法では直接的な影響と間接的な影響を区別して同定することが可能となり、転写制御の階層性を解明するための強力な手法といえます。
4.おわりに
全ての因子の影響が関与する細胞内における観察に加えて、転写制御因子の直接的な制御遺伝子を同定することにより、ゲノム転写制御のネットワークが明らかとなりつつあります。実際の細胞内におけるゲノム転写制御は動的であるため、ここにさらに時間経過における影響の観察を加えることにより、ゲノム転写制御システムの階層性の全体像が明らかになると考えています。今後は時間軸による観察も含めた解析を進めたいと考えています。
参考文献
1. Ishihama, A. Functional modulation of Escherichia coli RNA polymerase. Annu. Rev. Microbiol. 54, 499-518. (2000)
2. Martinez-Antonio, A. and Collado-Vides, J. Identifying global regulators in transcriptional regulatory networks in bacteria. Curr. Opin. Microbiol. 6, 482-9. (2003)
3. Shimada, T., Fujita, N., Maeda, M. and Ishihama, A. Systematic search for the Cra-binding promoters using genomic SELEX system. Genes Cells 10, 907-18.
4. Shimada, T., Yamamoto, K., Fujita, N. and Ishihama, A. Novel members of the cAMP receptor protein (CRP) regulon for transport and metabolism of carbon sources. PLoS ONE 6, e20081. (2011)
5. Shimada, T., Yamazaki, Y., Tanaka, K. and Ishihama, A. The whole set of constitutive promoters recognized by RNA polymerase RpoD holoenzyme of Escherichia coli. PLoS ONE 9, e90447. (2014)
6. Conway, T., Creecy, J. P., Maddox, S. M., Grissom, J. E., Conkle, T. L., Shadid, T. M., Teramoto, J., San, Miguel, P., Shimada, T., Ishihama, A., Mori, H. and Wanner, B. L. Unprecedented high-resolution view of bacterial operon architecture revealed by RNA sequencing. MBio. 5, e01442-14. (2014)



