RESEARCH_PROJECTS
細胞内レドックス状態可視化ツールの開発
タンパク質のレドックス(酸化還元)制御の研究を行う上で、研究対象のタンパク質が生体内でどのようなレドックス状態にあるのかを知ることは大変重要です。私達は、そのようなレドックス状態の「可視化」ツールの開発も行っています。
DNAマレイミド
タンパク質を構成するアミノ酸の一つであるシステインは、酸化還元の影響を受けやすく、かつタンパク質構造の決定に重要な分子内の共有結合の足場にもなっています。そのレドックス状態を知ることがタンパク質機能やその調節を明らかにする上で重要です。
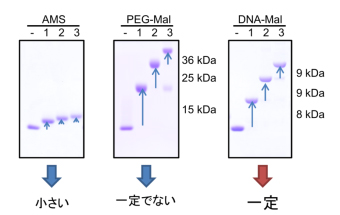
これまで、システインのチオール基のレドックス状態を探るツールとして、システインと特異的に反応するマレイミドをもつ化合物が用いられてきました。例えば、4-acetamido-4'-maleimidylstilbene(4?アセトアミド?4'?マレイミジルスチルベン=AMS)は、分子量540ほどでチオール基と特異的に反応し、タンパク質全体の分子量を約500大きくするので、この変化を電気泳動時のタンパク質の移動度変化として検出することが可能です。しかし、分子量が3万を超えるタンパク質の場合には、タンパク質そのものの分子量に比べて移動度の変化の割合が小さすぎて検出が困難でした。
この問題を克服するために、methoxypolyethylene glycol-maleimide(メトキシポリエチレングリコール・マレイミド=PEG マレイミド、通常使われるものは分子量約5,000)も用いられますが、PEG 分子の化学的な性質の制約から、電気泳動時に付加した分子量に見合った移動度の変化を示さないので、移動度の変化量から反応したシステインの数を知ることができませんでした。
AMSやPEGマレイミドのこれらの欠点を克服するために、私達はDNAを修飾剤として用いることにして、DNAマレイミドという化合物を開発しました。使用したのは24塩基の一本鎖DNAで、システインを修飾すると結合DNAの数の分だけ電気泳動の移動度を変化させることができます。結合したDNAあたりの移動度変化が分かっているので、タンパク質分子の中で還元状態にあるシステインのチオール基を、電気泳動の移動度変化から逆算して簡単に見積もることができるようになりました(図)。
■関連する最近の研究成果
-
Hara S, Nojima T, Seio K, Yoshida M, Hisabori T.

DNA-maleimide: an improved maleimide compound for electrophoresis-based titration of reactive thiols in a specific protein.
Biochim Biophys Acta. 2013 Apr;1830(4):3077-81. doi: 10.1016/j.bbagen.2013.01.012. Epub 2013 Jan 26. -
Hara S, Tatenaka Y, Ohuchi Y, Hisabori T.

Thioredoxin Selectivity for Thiol-based Redox Regulation of Target Proteins in Chloroplasts.
Biochem Biophys Res Commun. 2015 Jan 2;456(1):339-43. doi: 10.1016/j.bbrc.2014.11.082. Epub 2014 Nov 28.
レドックス感受性蛍光タンパク質
上記のように、DNAマレイミドなどの各種チオール修飾試薬を用いることで、生体内のタンパク質の酸化還元状態を可視化することができます。しかしながら、酸化還元状態の経時変化をより高い時間分解能で詳細に観察するためには、生きた細胞を用いてリアルタイムで酸化還元状態を測定する必要があります。これまでに、細胞内環境変化を生きたまま測定するための様々な蛍光センサータンパク質が作成され、細胞内のpHやCa2+濃度、ATP濃度などが測定されてきました。これらの蛍光センサータンパク質は主にオワンクラゲ由来の緑色蛍光タンパク質(GFP)を改変して作られています。酸化還元状態については、Hansonらが2005年に発表した酸化還元応答GFP(roGFP)を用いた研究が盛んに行われてきました。しかし、roGFPにも使い勝手の上でいくつか不都合な点があります。
私たちは、roGFPに変わる新たな酸化還元応答蛍光タンパク質センサーを作成し、光合成生物の細胞内酸化還元状態変化をより詳細に観察することを目標に研究を進めています。
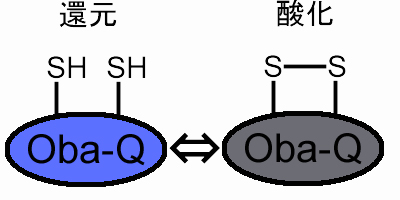
本プロジェクトではまず、群青色の蛍光を放つGFP変異体であるSiriusに対してアミノ酸変異を加えることで、酸化還元応答を示す新規の酸化還元応答タンパク質を作成しました。この新規の蛍光センサータンパク質は酸化される事によって蛍光が消えるという性質を持つことから、私たちはこのタンパク質をOba-Q (Oxidation balance sensed quenching proteins) と名づけました(下図)。
また、同様の手法を用いて水色の蛍光を放つシアン蛍光タンパク質(CFP)をベースとしたOba-Qcの開発にも成功しました。SiriusベースのOba-QsとCFPベースのOba-Qcとは励起・蛍光スペクトルが異なるため、細胞内に同時に発現させても互いのシグナルを容易に分けることができます。そのためroGFP単体では不可能であったオルガネラの多重染色などが可能となりました。高等植物では、葉緑体で光合成によって作られた過剰な還元力がミトコンドリアに輸送されるなど、様々なオルガネラが協調して酸化還元状態を維持していることが知られています。このようなオルガネラ間の酸化還元ネットワークの観察にOba-Qが大いに役立つと期待しています。

HeLa細胞内に発現させたOba-Qsの蛍光強度を共焦点蛍光顕微鏡を用いて観察しています。薬剤を用いて周囲の酸化還元状態を酸化側に偏らせることで蛍光が消え、再び還元することで蛍光が復活しているのが分かります。Oba-Qの蛍光強度を測定することでOba-Qがどの程度酸化されているかが分かり、そこから細胞内の酸化還元状態がどの程度酸化的なのかを計算することができます。
■関連する最近の研究成果
-
Sugiura K, Nagai T, Nakano M, Ichinose H, Nakabayashi T, Ohta N, Hisabori T.

Redox sensor proteins for highly sensitive direct imaging of intracellular redox state.
Biochem Biophys Res Commun. 2015 Feb 13;457(3):242-8. doi: 10.1016/j.bbrc.2014.12.095. Epub 2015 Jan 13.
